Back to top
Naturschutz Genetische Methoden

Genetische Methoden liefern neue Erkenntnisse über die Biologie der Arten sowie die räumliche und zeitliche Struktur der Populationen und werden daher für das Naturschutzmanagement immer bedeutender.
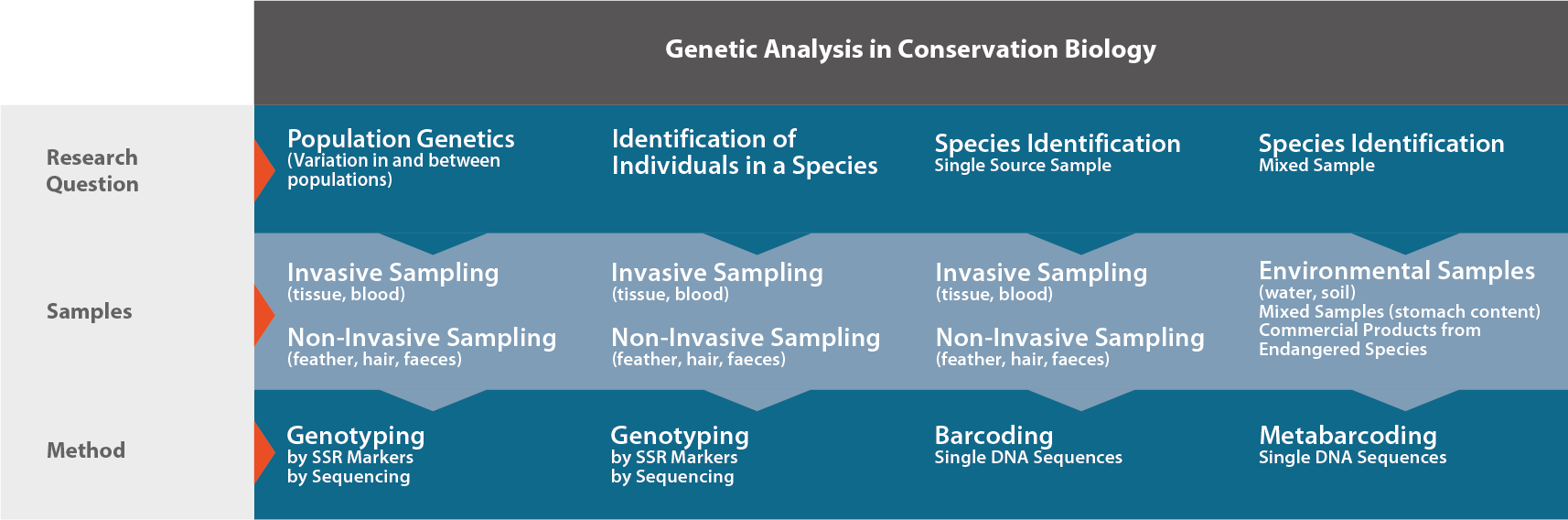
Jede naturschutzgenetische Studie wird durch Forschungsfragen initiiert, die von Experten des Naturschutzmanagements definiert werden. Je nach Fragestellung bietet die Naturschutzgenetik eine Vielzahl von Methoden, die wiederum das Versuchslayout, die statistische Analyse der Daten und die Empfehlungen für die Praxis definieren. Alle diese Schritte wie Probenahme, genetische Laboranalysen, statistische Auswertungen, Interpretation und Schlussfolgerungen müssen für jedes Projekt sorgfältig geplant werden. Konzentrieren Sie sich auf die praktischen Aspekte des Naturschutzes und überlassen Sie Ecogenics den kompletten Laborteil, einschließlich der Markerentwicklung, falls erforderlich, und der Genotypisierung von Proben. Darüber hinaus können wir Ihnen helfen, den Stichprobengrösse für Ihre Studie zu definieren und die aus der Studie gewonnenen Daten zu analysieren und zu interpretieren.
Jede naturschutzgenetische Studie wird durch Forschungsfragen initiiert, die von Experten des Naturschutzmanagements definiert werden. Je nach Fragestellung bietet die Naturschutzgenetik eine Vielzahl von Methoden, die wiederum das Versuchslayout, die statistische Analyse der Daten und die Empfehlungen für die Praxis definieren. Alle diese Schritte wie Probenahme, genetische Laboranalysen, statistische Auswertungen, Interpretation und Schlussfolgerungen müssen für jedes Projekt sorgfältig geplant werden. Konzentrieren Sie sich auf die praktischen Aspekte des Naturschutzes und überlassen Sie Ecogenics den kompletten Laborteil, einschließlich der Markerentwicklung, falls erforderlich, und der Genotypisierung von Proben. Darüber hinaus können wir Ihnen helfen, den Stichprobengrösse für Ihre Studie zu definieren und die aus der Studie gewonnenen Daten zu analysieren und zu interpretieren.
Übersicht
Methoden
Genotypisierung

Die biologische Vielfalt basiert auf drei Ebenen. Grundlage ist die genetische Vielfalt, gefolgt von der Artenvielfalt und der Vielfalt der Ökosysteme. Durch die Genotypisierung von Individuen einer einzelnen Art, die aus verschiedenen Populationen gesammelt wurden, sind Einblicke in die genetische Vielfalt möglich, so dass Rückschlüsse auf das Verhalten der Art gezogen werden können.
Die am häufigsten verwendeten Marker zur Untersuchung genetischer Variationen sind Mikrosatellitenmarker (SSR: simple sequence repeat). Mikrosatelliten-Marker sind artspezifisch, von beiden Elternteilen vererbt (co-dominante Vererbung), selektiv neutral und normalerweise hochpolymorph. Durch die Genotypisierung mehrerer verschiedener Mikrosatellitenmarker zeigt jedes Individuum ein ausgeprägtes allelisches Muster, das es erlaubt, Individuen zu identifizieren und die Populationsgröße zu bestimmen.
Darüber hinaus können die Häufigkeiten von Allelen zwischen Individuen, die aus verschiedenen Populationen gesammelt wurden, verwendet werden, um populationsgenetische Maßnahmen wie die genetische Variation in und zwischen Populationen zu berechnen und Rückschlüsse auf die genetische Struktur der Population, Inzucht, Hybridisierung und Genfluss zu ziehen. Der Genfluss ist ein Maß für den genetischen Austausch zwischen Populationen und ermöglicht es, die Frage nach der Vernetzung von Populationen zu beantworten. Darüber hinaus kann durch die Analyse der Populationsstruktur zu verschiedenen Zeitpunkten das Ausmaß der genetischen Vielfalt und ihre Veränderung im Laufe der Zeit untersucht werden. Die Analyse der Populationsstruktur vor und nach Maßnahmen des Naturschutzmanagements zur Reduzierung der Lebensraumfragmentierung kann beispielsweise für bestimmte Arten untersucht werden, um festzustellen, ob die getroffenen Maßnahmen erfolgreich waren.
Kleine Mengen an DNA reichen bereits aus, um eine Genotypisierung durch Mikrosatelliten durchzuführen. Ecogenics hat ausserdem die Expertise in der DNA-Isolierung von nicht-invasiven Proben (Stuhl, Haare, Federn, Eierschale, etc.). Diese Methode eignet sich auch für verschiedene Arten, die schwer zu beobachten sind. So kann beispielsweise die Anzahl der Individuen in gefährdeten Arten wie Tetrao urogallus (Auerhuhn) ohne invasive Probenahme geschätzt und die daraus resultierenden Daten für die Abstammungsanalyse genutzt werden.
Um höchste Datenqualität zu gewährleisten und den Fortschritt Ihrer Projekte zu beschleunigen, bietet ecogenics das gesamte Spektrum der Genotypisierung an. Daher führen wir gerne Ihre Genotypisierungsprojekte durch, die auf von der ecogenics entwickelten Markern oder auf Markern aus anderen Quellen (intern, veröffentlicht etc.) basieren. Unsere langjährige Erfahrung mit der DNA-Isolierung aus vielen Quellen, kombiniert mit unserer Expertise in der PCR und der Fragmentlängenanalyse, garantiert optimale Ergebnisse. Ob Sie fluoreszierende Tracefiles (.fsa), eine Alleltabelle mit allen identifizierten Allelen pro Marker und Probe oder weitere bioinformatische Analysen erhalten möchten, liegt bei Ihnen.
Zögern Sie nicht, uns zu kontaktieren, wir besprechen gerne Ihr geplantes Projekt und erstellen Ihnen ein individuelles Angebot.
Die am häufigsten verwendeten Marker zur Untersuchung genetischer Variationen sind Mikrosatellitenmarker (SSR: simple sequence repeat). Mikrosatelliten-Marker sind artspezifisch, von beiden Elternteilen vererbt (co-dominante Vererbung), selektiv neutral und normalerweise hochpolymorph. Durch die Genotypisierung mehrerer verschiedener Mikrosatellitenmarker zeigt jedes Individuum ein ausgeprägtes allelisches Muster, das es erlaubt, Individuen zu identifizieren und die Populationsgröße zu bestimmen.
Darüber hinaus können die Häufigkeiten von Allelen zwischen Individuen, die aus verschiedenen Populationen gesammelt wurden, verwendet werden, um populationsgenetische Maßnahmen wie die genetische Variation in und zwischen Populationen zu berechnen und Rückschlüsse auf die genetische Struktur der Population, Inzucht, Hybridisierung und Genfluss zu ziehen. Der Genfluss ist ein Maß für den genetischen Austausch zwischen Populationen und ermöglicht es, die Frage nach der Vernetzung von Populationen zu beantworten. Darüber hinaus kann durch die Analyse der Populationsstruktur zu verschiedenen Zeitpunkten das Ausmaß der genetischen Vielfalt und ihre Veränderung im Laufe der Zeit untersucht werden. Die Analyse der Populationsstruktur vor und nach Maßnahmen des Naturschutzmanagements zur Reduzierung der Lebensraumfragmentierung kann beispielsweise für bestimmte Arten untersucht werden, um festzustellen, ob die getroffenen Maßnahmen erfolgreich waren.
Kleine Mengen an DNA reichen bereits aus, um eine Genotypisierung durch Mikrosatelliten durchzuführen. Ecogenics hat ausserdem die Expertise in der DNA-Isolierung von nicht-invasiven Proben (Stuhl, Haare, Federn, Eierschale, etc.). Diese Methode eignet sich auch für verschiedene Arten, die schwer zu beobachten sind. So kann beispielsweise die Anzahl der Individuen in gefährdeten Arten wie Tetrao urogallus (Auerhuhn) ohne invasive Probenahme geschätzt und die daraus resultierenden Daten für die Abstammungsanalyse genutzt werden.
Um höchste Datenqualität zu gewährleisten und den Fortschritt Ihrer Projekte zu beschleunigen, bietet ecogenics das gesamte Spektrum der Genotypisierung an. Daher führen wir gerne Ihre Genotypisierungsprojekte durch, die auf von der ecogenics entwickelten Markern oder auf Markern aus anderen Quellen (intern, veröffentlicht etc.) basieren. Unsere langjährige Erfahrung mit der DNA-Isolierung aus vielen Quellen, kombiniert mit unserer Expertise in der PCR und der Fragmentlängenanalyse, garantiert optimale Ergebnisse. Ob Sie fluoreszierende Tracefiles (.fsa), eine Alleltabelle mit allen identifizierten Allelen pro Marker und Probe oder weitere bioinformatische Analysen erhalten möchten, liegt bei Ihnen.
Zögern Sie nicht, uns zu kontaktieren, wir besprechen gerne Ihr geplantes Projekt und erstellen Ihnen ein individuelles Angebot.
Barcoding

Die Klassifizierung von Arten wird oft dadurch erschwert, dass die Struktur für eine morphologische Klassifikation wie z.B. reproduktive Strukturen in Pflanzen oder Pilzen nicht vorhanden ist. Darüber hinaus sind viele Arten aufgrund winziger morphologischer Unterschiede, die ein fundiertes Expertenwissen erfordern, schwer zu klassifizieren. DNA-basiertes Barcoding kann helfen, einige der Einschränkungen der klassischen Klassifizierung von Arten zu überwinden. Die Forschungsgemeinschaft investierte erhebliche Anstrengungen, um DNA-basierte Marker für die Klassifizierung molekularer Arten zu finden. Die genetische Identifizierung einer Art basiert auf der Analyse einer kurzen DNA-Sequenz (hauptsächlich aus Mitochondrien und Chloroplasten oder ribosomaler DNA), die mit einer Referenzdatenbank verglichen werden kann.
ecogenics bietet einen Full-Service von der DNA-Isolierung bis zur Artenidentifikation für alle Organismengruppen. Unser Qualitätsservice umfasst die DNA-Isolierung aus vielen Ausgangsmaterialien (Gewebe, Blut, Haare, Kot, Federn, Eierschalen, etc.), gefolgt von der PCR-Amplifikation des entsprechenden Barcode-Locus für die jeweilige Organismengruppe und der Sanger-Sequenzierung. Abschließend identifizieren wir die Arten auf der Grundlage öffentlich zugänglicher Datenbanken und stellen Ihnen ein Zertifikat zur Identifizierung der Arten zur Verfügung.
ecogenics bietet einen Full-Service von der DNA-Isolierung bis zur Artenidentifikation für alle Organismengruppen. Unser Qualitätsservice umfasst die DNA-Isolierung aus vielen Ausgangsmaterialien (Gewebe, Blut, Haare, Kot, Federn, Eierschalen, etc.), gefolgt von der PCR-Amplifikation des entsprechenden Barcode-Locus für die jeweilige Organismengruppe und der Sanger-Sequenzierung. Abschließend identifizieren wir die Arten auf der Grundlage öffentlich zugänglicher Datenbanken und stellen Ihnen ein Zertifikat zur Identifizierung der Arten zur Verfügung.
Metabarcoding

Das Metabarcoding ist eine schnelle Methode zur Beurteilung der Biodiversität, die den Einsatz von DNA-basierter Identifizierung (Barcoding) und Hochdurchsatz-DNA-Sequenzierung kombiniert. Dies ermöglicht es, das Vorhandensein von Gemeinschaften innerhalb einer Probe aus winzigen Mengen an DNA zu beurteilen, die aus Umweltproben wie Stuhl, Wasser oder Boden isoliert wurden. Eine klassische Anwendung des Metabarcodings ist die Untersuchung von Bakterien- und Pilzgemeinschaften. Aber der gleiche Ansatz kann auch für nahezu jede beliebige Organismengruppe angewendet werden, z.B. für den Nachweis von amphibischer DNA in Wasserproben oder für die Analyse von Tierfutter.
Abhängig vom Resultat der Metabarcoding-Analyse ist die Verfügbarkeit einer gut gepflegten Referenzdatenbank, um die bei der Analyse der Proben erzeugten Sequenzen mit der Taxonomie zu verknüpfen. Obwohl für einige Organismengruppen viele öffentliche Referenzdatenbanken zur Verfügung stehen, kann es in einigen Fällen auch sinnvoll sein, eine eigene Referenzdatenbank mit gut charakterisierten Proben aufzubauen.
Ecogenics entwickelte und validierte verschiedene Metabarcoding-Anwendungen und optimierte die bioinformatische Analyse, um die NGS Sequenzierdaten zu analysieren. Unser Qualitätsservice umfasst die DNA-Isolierung aus vielen Quellen (Wasser, Stuhl, Boden, etc.), gefolgt von der PCR-Amplifikation des geeigneten Barcode-Locus für das Metabarcoding mit Universalprimern, der Next-Generation Sequenzierung sowie der bioinformatischen Analyse.
Abhängig vom Resultat der Metabarcoding-Analyse ist die Verfügbarkeit einer gut gepflegten Referenzdatenbank, um die bei der Analyse der Proben erzeugten Sequenzen mit der Taxonomie zu verknüpfen. Obwohl für einige Organismengruppen viele öffentliche Referenzdatenbanken zur Verfügung stehen, kann es in einigen Fällen auch sinnvoll sein, eine eigene Referenzdatenbank mit gut charakterisierten Proben aufzubauen.
Ecogenics entwickelte und validierte verschiedene Metabarcoding-Anwendungen und optimierte die bioinformatische Analyse, um die NGS Sequenzierdaten zu analysieren. Unser Qualitätsservice umfasst die DNA-Isolierung aus vielen Quellen (Wasser, Stuhl, Boden, etc.), gefolgt von der PCR-Amplifikation des geeigneten Barcode-Locus für das Metabarcoding mit Universalprimern, der Next-Generation Sequenzierung sowie der bioinformatischen Analyse.
Marker Systeme
Verfügbare Marker Systeme für:
- Genotypisierung Indikatorarten
Die Vernetzung von Biotopen ist eine wichtige Naturschutzstrategie in der Schweiz. Indikatoren für die Vernetzung und/oder Fragmentierung von Biotopen sind weit verbreitete, mittelstarke Arten mit einer führenden Artenfunktion für Lebensräume. Ecogenics bietet molekulargenetische Analysen für die folgenden Indikatorarten an:
Melittis melissophyllum (Immenblatt)
Stethophyma grossum (Sumpfschrecke)
Metrioptera bicolor (= Bicolorana bicolor) Zweifarbige Beisschrecke
Zebrina detrita (Zebraschnecke, Weisse Turmschnecke)
Bombina variegata variegata (Gelbbauchunke)
Triturus cristatus & Triturus carnifex
- Metabarcoding: Amphibiennachweis
Amphibien haben sowohl im Biotop- als auch im Artenschutz einen besonders hohen Stellenwert. Für das Monitoring von Amphibien in aquatischen Ökosystemen bietet ecogenics den Nachweis und die Identifizierung von Amphibien auf der Grundlage molekulargenetischer Analysen mittels eDNA (Wasserprobe) Metabarcoding an.
- Barcoding: BOLDSYSTEMS
Unsere molekulargenetischen Analysen mit universellen Markersystemen ermöglichen die Identifizierung von Arten mit dem Barcode of Life Data System (bold), einer Informatik-Workbench, die die Veröffentlichung von DNA-Barcode-Datensätzen unterstützt.
Tieridentifizierung (COI)
Pilzidentifizierung (ITS)
Pflanzenidentifizierung (RBCL & MATK)
Wenn Sie noch kein geeignetes Markersystem für Ihre Art oder Forschungsfrage gefunden haben, zögern Sie bitte nicht, uns zu kontaktieren, Markersysteme wurden bereits für viele Arten entwickelt und Details finden Sie oft in der Literatur. Darüber hinaus ist ecogenics auf die Entwicklung von Markersystemen spezialisiert und wir würden uns freuen, mit Ihnen an Ihrem Projekt zu arbeiten. Nehmen Sie Kontakt mit uns auf, um zu sehen, wie wir Ihre Forschung bereichern können.