
Back to top
Genotypisierung durch SSRs

Um höchste Datenqualität zu gewährleisten und den Fortschritt Ihrer Forschungsprojekte zu beschleunigen, bietet ecogenics das gesamte Spektrum der Genotypisierung an. Gerne führen wir Ihre Genotypisierungsprojekte durch. Dabei kommt es nicht darauf an ob die Marker bereits von ecogenics entwickelt wurden oder ob es sich um Marker aus diversen anderen Quellen (intern, veröffentlicht etc.) handelt. Unsere langjährige Erfahrung mit der DNA-Isolierung aus verschiedenen Ursprungsmaterialien, kombiniert mit unserer PCR -Expertise, garantiert optimale Ergebnisse in kurzer Zeit (6 bis 10 Wochen, je nach Projektgröße).
Workflow
Ein typischer Workflow für eine Genotypisierung mit SSR Markern ist in der folgenden Abbildung dargestellt. Bitte beachten Sie, dass unser modularer Genotypisierungsprozess verschiedene Ein- und Ausstiegsoptionen ermöglicht. Ob Sie die gesamte Genotypisierung oder nur Teile davon auslagern möchten, können Sie frei entscheiden.

Produkte
SSR Multiplexing
Für ein Genotypisierungsprojekt mit einer größeren Anzahl von Proben und Markern sind Multiplex-PCR-Reaktionen gegenüber Singleplex-Reaktionen kostengünstiger. Aus diesem Grund bietet Ihnen ecogenics das Multiplexen von SSRs Service an. Je nach Anzahl der Marker und Proben empfehlen wir die Verwendung von 3 bis 7-plex PCRs. Die SSR-Marker Information kann entweder aus einem ecogenics SSR-Entwicklungsprojekt oder aus anderen Quellen (intern, veröffentlicht, etc) stammen.
Standardmäßig verwenden wir die Fluoreszenzfarbstoffe FAM, ATTO532, ATTO550 und ATTO565, die mit ABIs Standardfilterset G5 kompatibel sind. Neben einer detaillierten Assaybeschreibung erhalten die Kunden 100 µM Stammlösungen aller im Assay verwendeten Primer, einschließlich farbstoffmarkierter Vorwärtsprimer.
Standardmäßig verwenden wir die Fluoreszenzfarbstoffe FAM, ATTO532, ATTO550 und ATTO565, die mit ABIs Standardfilterset G5 kompatibel sind. Neben einer detaillierten Assaybeschreibung erhalten die Kunden 100 µM Stammlösungen aller im Assay verwendeten Primer, einschließlich farbstoffmarkierter Vorwärtsprimer.
Publizierte SSR-Markerprüfung
Falls SSR Marker bereits für Ihre Spezies entwickelt wurden und Sie sich nicht sicher sind, ob diese veröffentlichten Marker stabil funktionieren, prüfen wir die Funktionalität und Polymorphie für jeden Locus in 7 Individuen für Sie.
Neben einer detaillierten Assaybeschreibung erhalten unsere Kunden ebenfalls die 100 µM Primer Stammlösungen der getesteten Loci, wobei die Vorwärtsprimer mit einem FAM-Tag gelabelt sind.
Neben einer detaillierten Assaybeschreibung erhalten unsere Kunden ebenfalls die 100 µM Primer Stammlösungen der getesteten Loci, wobei die Vorwärtsprimer mit einem FAM-Tag gelabelt sind.
PCR & Kapillarelektrophorese
Das Knowhow der ecogenics im Bereich der PCR und Kapillarelektrophorese ist sehr gross. Unsere Prozesse sind optimiert und automatisiert und durch unsere hohen Standards gewährleisten wir zuverlässige Daten.
Für die Kapillarelektrophorese verwenden wir die Applied Biosystems 3730xl DNA Analyzer Plattform. Standardmäßig verwenden wir die Fluoreszenzfarbstoffe FAM, ATTO532, ATTO550 und ATTO565, die mit ABIs Standardfilterset G5 kompatibel sind.
Ob Sie fluoreszierende Trace-Dateien (.fsa) oder auch eine Alleltabelle mit allen identifizierten Allelen pro Marker und Probe erhalten möchten, liegt bei Ihnen.
Genotype Calling
Mit unserem Analyseservice werden fluoreszierende Trace-Dateien (.fsa) dem Analysetool SoftGenetics GeneMarker® automatisierten Analyseverfahren unterzogen. Nach Abschluss der Analyse mit GeneMarker® wird die Gültigkeit jedes Allels durch eine visuelle Inspektion überprüft, was eine hohe Genauigkeit der gelieferten Ergebnisse garantiert. Im Lieferumfang enthalten ist eine Alleltabelle (.xlsx) mit allen identifizierten Allelen pro Marker und Probe.
Resultate
Die Durchführung des SSR-Genotypisierungsprojekts umfasst:
- eine Zusammenfassung des Genotypisierungsprojekts
- die Elektropherogramme als Rohdaten im ABI-Format (.fsa)
- Stammlösungen der gelabelten und ungelabelten Primer (100 µM)
- Wurde vor der Genotypisierung ein SSR-Multiplexing durchgeführt, gehört eine Read&Go-Broschüre mit detaillierten Informationen über die multiplexierte PCR, die entsprechenden PCR-Protokolle und ein Bild des Elektropherogramms (siehe Abbildung 1) zum Lieferumfang.
- Wenn Sie sich zusätzlich für das Produkt Genotype Calling entschieden haben, ist eine Excel-Datei (siehe Abbildung 2) mit den Allelgrößen pro Locus und Probe im Lieferumfang enthalten.
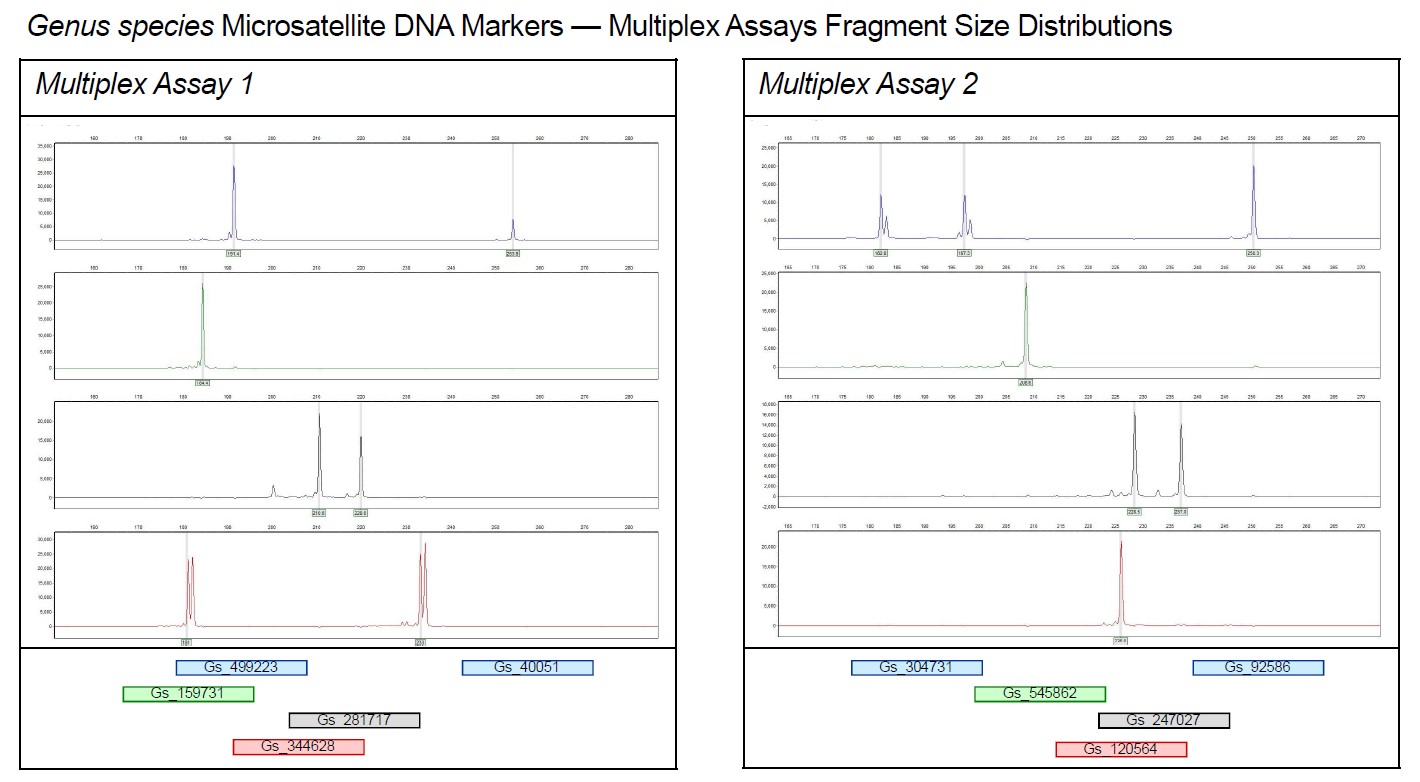
Abbildung 1: Beispiele für Elektropherogramme von 2 multiplexierten PCRs für ein einzelnes Individuum der Zielspezies. Die Markierung der Fragmente mit verschiedenen Farbstoffen ermöglicht die eindeutige Erkennung der einzelnen Loci innerhalb der Multiplex-PCRs.
Abbildung 2: Beispiel für einen Allelbericht im Rahmen des SSR-Genotypisierungsprojekts. Für jede Probe werden die an jedem Locus gefundenen Allele aufgelistet, so dass der Allelbericht für jede nachfolgende Analyse in Populationsgenetik-Software (d.h. Struktur, Arlequin, ....) neu formatiert werden kann.
