
Back to top
Genotyping-by-Sequencing (GBS)

Mit dem rasanten Fortschritt der Sequenzierungstechnologien der nächsten Generation wurde die Genotypisierung durch Sequenzierung zu einem sehr attraktiven Preis möglich. Im Gegensatz zu herkömmlichen Genotypisierungsmethoden erfordert die SNP (Single Nucleotide Polymorphism) Genotypisierung durch Sequenzierung der nächsten Generation keine vorherige Markerentwicklung oder Sequenzinformation, sondern ermöglicht eine direkte SNP-Genotypisierung jeder Population von Interesse.
Wir freuen uns, Ihr Projekt mit dieser bahnbrechenden Technologie zu unterstützen, bitte zögern Sie nicht, uns zu kontaktieren, um mögliche Projektlayouts zu besprechen.
Wir freuen uns, Ihr Projekt mit dieser bahnbrechenden Technologie zu unterstützen, bitte zögern Sie nicht, uns zu kontaktieren, um mögliche Projektlayouts zu besprechen.
Workflow
Ein typischer Workflow für ein Genotyping-by-Sequencing-Projekt ist in der folgenden Abbildung dargestellt. Bitte beachten Sie, dass unser modularer Genotypisierungsprozess verschiedene Ein- und Ausstiegsoptionen ermöglicht. Ob Sie das gesamte Genotypisierungsprojekt oder nur Teile davon auslagern möchten, liegt ganz bei Ihnen.
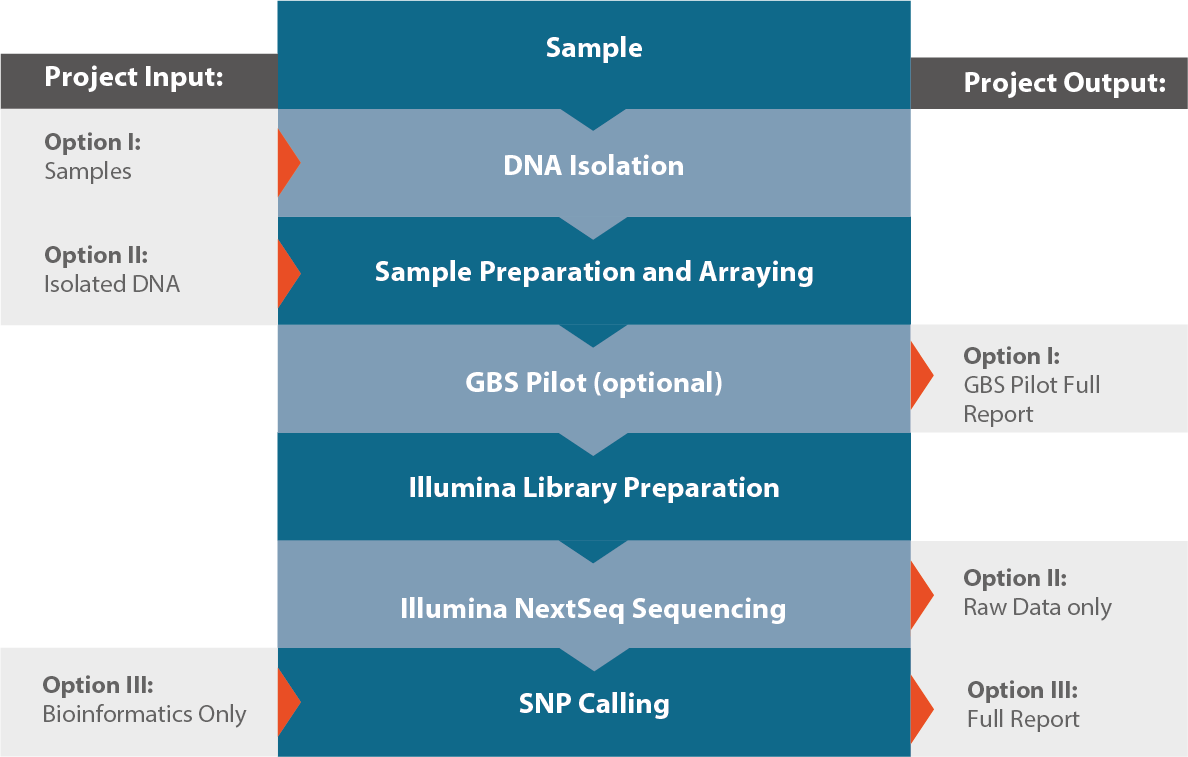
Produkt
GBS Pilot
Mit dem GBS Pilot bieten wir Ihnen die Möglichkeit, das GBS-Protokoll für Ihre Spezies auf einer kleinen Probenmenge (5-10 Proben) zu testen und zu optimieren. Dieser Service umfasst die Erstellung von individuell mit MID-Tags versehenen, reduzierten Libraries, die Sequenzierung auf der Illumina MiSeq-Plattform und die bioinformatische Analyse. Diese Option ist besonders vorteilhaft für mittelgrosse Projekte oder für die Methodenvalidierung.
GBS Batch-Lauf
Dieser Service umfasst die Erstellung von individuell MID-getaggten, reduzierten Libraries und die Sequenzierung auf einer Illumina NextSeq-Plattform. Abhängig von Ihren Zielen bieten wir die Sequenzierung verschiedener Batchgrößen, sowie die Sequenzierung von Leselängen an. Die GBS Batch Runs sind sinnvoll, wenn größere Chargen von Proben parallel analysiert werden können.
Bioinformatik
Da die Analyse großer und komplexer GBS-Datensätze einige spezifische Kenntnisse in der Bioinformatik erfordert, können wir die Basisanalyse durchführen, um SNPs im Datensatz zu identifizieren. Die Analyse umfasst das Clustering von sehr ähnlichen Sequenzen für jede Probe (z.B. Stacks), die Identifizierung von SNPs innerhalb der Proben, sowie die Identifizierung von SNP-Loci über die in der Studie enthaltenen Proben (z.B. Katalogaufbau). Das Ergebnis ist eine Tabelle mit dem SNP-Aufruf pro Locus und Probe.
Falls für Ihre Spezies ein Referenzgenom verfügbar ist, wird der Stack auf das Genom abgebildet, so dass Sie zusätzliche Informationen über den Standort der einzelnen SNPs erhalten.
Das Standardformat unserer Lieferung ist das VCF (Variant Call Format). Falls Sie ein bestimmtes Dateiformat für weitere Analysen benötigen, kontaktieren Sie uns und wir werden eine Lösung für Sie finden.
Falls für Ihre Spezies ein Referenzgenom verfügbar ist, wird der Stack auf das Genom abgebildet, so dass Sie zusätzliche Informationen über den Standort der einzelnen SNPs erhalten.
Das Standardformat unserer Lieferung ist das VCF (Variant Call Format). Falls Sie ein bestimmtes Dateiformat für weitere Analysen benötigen, kontaktieren Sie uns und wir werden eine Lösung für Sie finden.
Resultate
Die Lieferung eines Genotyping-by-Sequencing-Projekts umfasst:
- eine Zusammenfassung des GBS-Projekts
- die Rohdaten (Fastq-Dateien)
Falls wir die Sequenz-Assemblierung und -Filterung für SNP-Kandidaten mit dem STACKS-Paket durchführen, erhalten Sie zusätzlich eine Tabelle mit dem SNP-Aufruf pro Locus und Sample (Abbildung 1). Darüber hinaus werden, falls vorhanden, die Informationen eines Referenzgenoms in die Analyse einbezogen (Abbildung 2).
Abbildung 1. Genotyp-Tabelle mit SNP-Aufrufen pro Locus und Probe für eine genombasierte Referenz-Analysestrategie.
Abbildung 2. Schematische Darstellung von GBS-Datenanalysestrategien. Falls ein Referenzgenom verfügbar ist, werden die aus den reduzierten Repräsentationsbibliotheken gewonnenen Sequenzen gegen das Referenzgenom abgebildet und die SNPs abgefragt, was zu einer Genotypentabelle inkl. der Koordinate des SNP auf dem Referenzgenom führt. Diese Informationen sind besonders hilfreich, wenn PCR-basierte Assays für einen begrenzten Satz von SNPs entwickelt werden sollen. Im Gegensatz dazu kann für alle Nicht-Modellorganismen eine De-Novo-Analysestrategie angewendet werden, die zu einer Genotyp-Tabelle führt. In der De-novo-Analysestrategie sind jedoch keine Informationen über die Standorte der SNPs verfügbar.
